



RNA結合蛋白免疫共沉淀RIP-seq
RNA結合蛋白(RNA binding protiens)結合RNA調控基因表達,與RNA相互作用是聯系多組學的橋梁。從表觀遺傳到蛋白代謝,RNA蛋白互作的研究是高分文章必不可少的。產品介紹經典案例結果展示送樣建議RNA結合蛋白免疫共沉淀測序(RIP-seq)
—全轉錄組范圍揭示RNA與蛋白互作技術
RNA結合蛋白(RNA binding protiens)結合RNA調控基因表達,與RNA相互作用是聯系多組學的橋梁。從表觀遺傳到蛋白代謝,RNA蛋白互作的研究是高分文章必不可少的。RIP-seq(RNA Binding Protein Immunoprecipitation Assay Sequencing,RNA 結合蛋白免疫沉淀測序)通過特定抗體把相應的RNA-蛋白復合物富集沉淀下來,經過分離純化就可以對結合該區域RNA序列進行高通量測序,在轉錄組范圍內鑒定與蛋白結合的區域,是了解轉錄后調控網絡的有力工具。為多組學助力機制研究,艾斯基因引入RIP-seq這一研究轉錄后動態調控技術,精確定位RNA與蛋白互作,助力廣大科研工作者發表高分SCI文章!
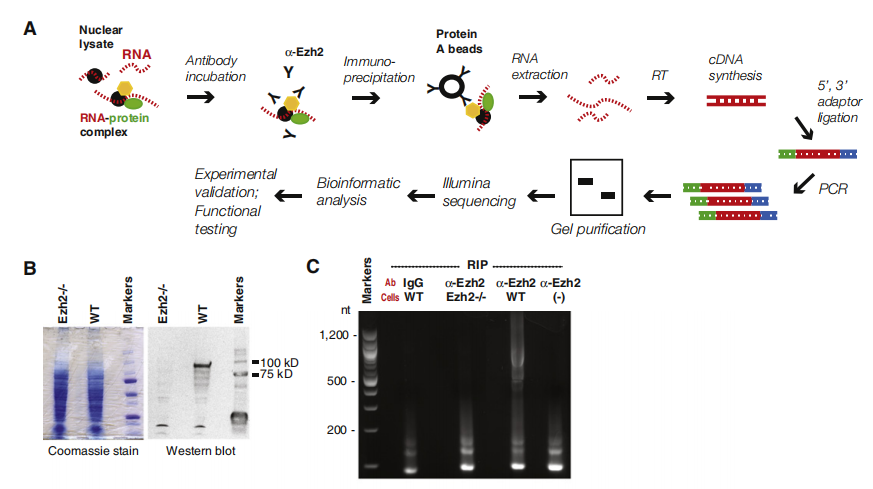
RIP-seq實驗原理圖
艾斯優勢
項目經驗豐富
已經完成人、動植物等幾十個物種, 5000+例樣本項目經驗
專業生信分析團隊
針對多組學測序,艾斯搭建多組學關聯分析流程,多維度助力科研機制研究
高效自動化流程
自動化樣本處理、建庫及分析流程,保障高效周期,40天極速交付
高水平文章經驗
團隊已經發表Genome Biol、Nat Commun、Cell Res等高水平SCI文章
樣本類型和送樣要求
樣本類型
送樣建議
新鮮動物組織
>50 mg
新鮮植物組織
>300 mg
細胞樣本
>5×106個
備注:詳細送樣要求參考 RIP-seq送樣要求2024
數據信息分析
ATAC-seq
分析內容
備注
標準分析
1、測序數據質量評估
過濾掉低質量數據,保證數據質量
2、與參考基因組比對
比對率和覆蓋度分析
3、基因組 Peak 分析
全轉錄組范圍RBP結合RNA的Peak特征
4、Peak關聯基因的功能分析
Peak所關聯基因的GO和KEGG功能富集
5、組間差異Peak分析
尋找差異Peak及注釋
6、組間差異Peak功能分析
差異Peak的GO,KEGG功能富集分析
關聯分析
7、甲基化組學關聯分析
不同甲基化水平基因、DMR的Peak信號分布等
8、轉錄組關聯分析
不同表達水平基因、DEG的Peak信號分布等
9、其它定制化分析
結合課題背景亮點挖掘
在研究中大多數技術都不是單一使用,RIP-seq也不例外,它會搭配其他組學一起對研究的深度和廣度進行擴充。RIP-seq可以與多種技術聯合分析,RNA-seq、m6A-seq等。
案例分析1:m6A識別蛋白YTHDF1在低氧適應和非小細胞肺癌發生發展中的重要功能
YTHDF1 links hypoxia adaptation and non-small cell lung cancer progression. Nat Commun 10, 4892 (2019)..
研究內容:研究發現YTHDF1作為m6A修飾后的RNA結合蛋白家族成員之一,在高原家養哺乳動物中低表達,而在正常肺上皮細胞中抑制其表達可以抵抗低氧誘導的細胞凋亡。深入研究發現,YTHDF1在非小細胞肺癌腫瘤組織和細胞系中均高表達,其在常氧條件下通過加速CDK2和CDK4等細胞周期蛋白的表達來促進腫瘤細胞的增殖;而在鉑類藥物為主的化療壓力環境下,由于YTHDF1高表達促進了Keap1蛋白的表達,導致Nrf2轉錄因子的迅速降解和下游耐藥基因AKR1C1的沉默,因此腫瘤患者對化療更敏感且總生存時間更長
研究技術:RIP-seq、m6A-seq、RNA-seq
研究總結:聯合運用了RIP-seq和m6A-seq,鑒定出細胞周期因子CDK2和CDK4的m6A修飾發生了變化,且被YTHDF1所識別和結合。CDK2和CDK4表達的紊亂也導致細胞周期的紊亂,使得癌細胞增殖加速
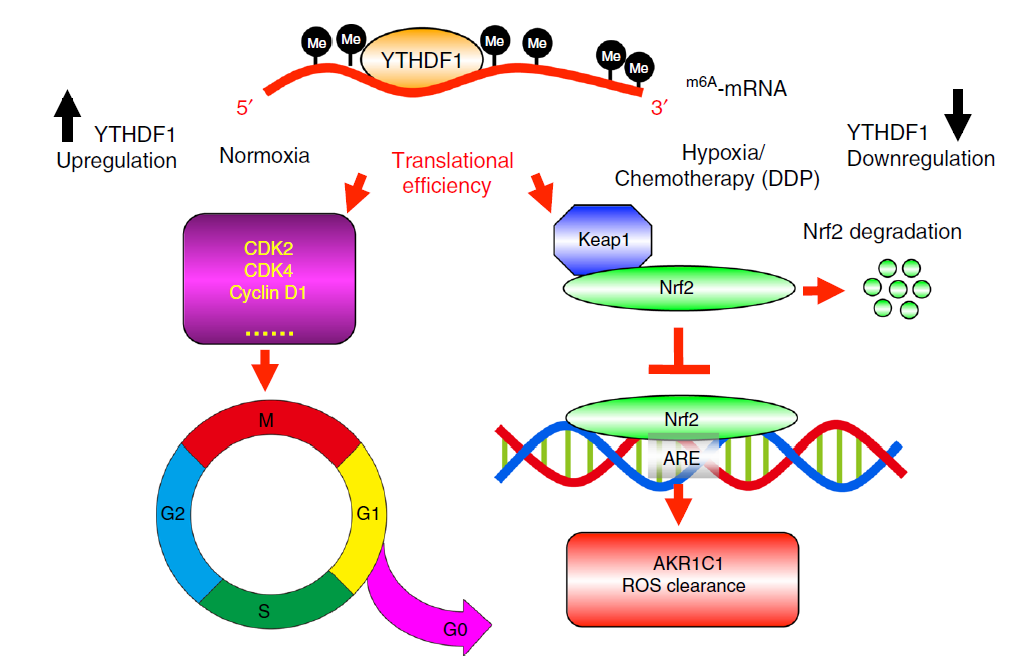
圖1 m6A-seq和 RIP-seq揭示YTHDF1調控模型
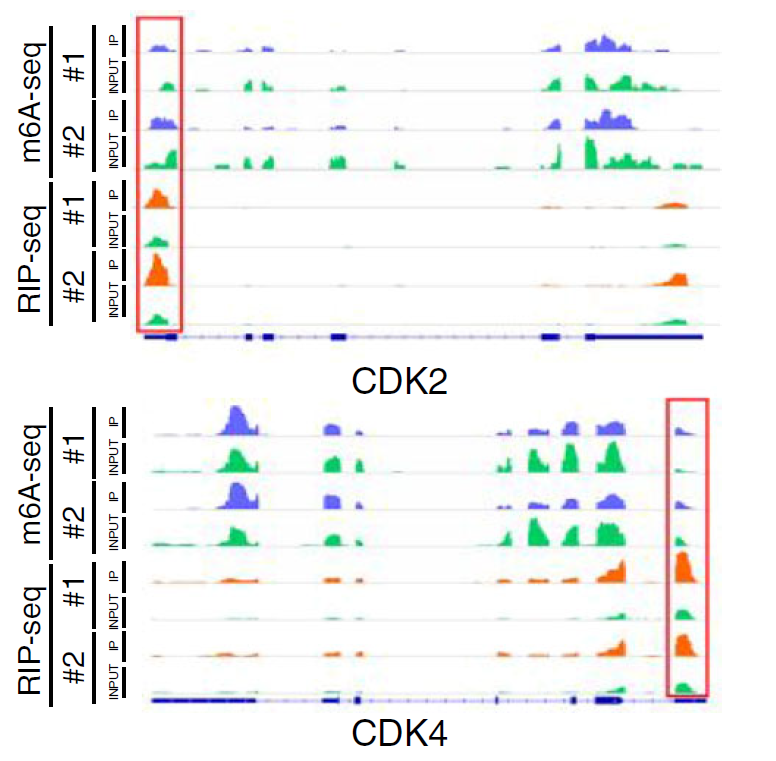
圖2 IGV圖展示CDK2和CDK4的RNA結合蛋白富集差異和m6A修飾差異
案例內容
結果內容
建議內容

RNA結合蛋白免疫共沉淀RIP-seq
RNA結合蛋白(RNA binding protiens)結合RNA調控基因表達,與RNA相互作用是聯系多組學的橋梁。從表觀遺傳到蛋白代謝,RNA蛋白互作的研究是高分文章必不可少的。0755-28507994產品介紹經典案例結果展示送樣建議RNA結合蛋白免疫共沉淀測序(RIP-seq)
—全轉錄組范圍揭示RNA與蛋白互作技術
RNA結合蛋白(RNA binding protiens)結合RNA調控基因表達,與RNA相互作用是聯系多組學的橋梁。從表觀遺傳到蛋白代謝,RNA蛋白互作的研究是高分文章必不可少的。RIP-seq(RNA Binding Protein Immunoprecipitation Assay Sequencing,RNA 結合蛋白免疫沉淀測序)通過特定抗體把相應的RNA-蛋白復合物富集沉淀下來,經過分離純化就可以對結合該區域RNA序列進行高通量測序,在轉錄組范圍內鑒定與蛋白結合的區域,是了解轉錄后調控網絡的有力工具。為多組學助力機制研究,艾斯基因引入RIP-seq這一研究轉錄后動態調控技術,精確定位RNA與蛋白互作,助力廣大科研工作者發表高分SCI文章!

RIP-seq實驗原理圖
艾斯優勢
項目經驗豐富
已經完成人、動植物等幾十個物種, 5000+例樣本項目經驗
專業生信分析團隊
針對多組學測序,艾斯搭建多組學關聯分析流程,多維度助力科研機制研究
高效自動化流程
自動化樣本處理、建庫及分析流程,保障高效周期,40天極速交付
高水平文章經驗
團隊已經發表Genome Biol、Nat Commun、Cell Res等高水平SCI文章
樣本類型和送樣要求
樣本類型
送樣建議
新鮮動物組織
>50 mg
新鮮植物組織
>300 mg
細胞樣本
>5×106個
備注:詳細送樣要求參考 RIP-seq送樣要求2024
數據信息分析
ATAC-seq
分析內容
備注
標準分析
1、測序數據質量評估
過濾掉低質量數據,保證數據質量
2、與參考基因組比對
比對率和覆蓋度分析
3、基因組 Peak 分析
全轉錄組范圍RBP結合RNA的Peak特征
4、Peak關聯基因的功能分析
Peak所關聯基因的GO和KEGG功能富集
5、組間差異Peak分析
尋找差異Peak及注釋
6、組間差異Peak功能分析
差異Peak的GO,KEGG功能富集分析
關聯分析
7、甲基化組學關聯分析
不同甲基化水平基因、DMR的Peak信號分布等
8、轉錄組關聯分析
不同表達水平基因、DEG的Peak信號分布等
9、其它定制化分析
結合課題背景亮點挖掘
在研究中大多數技術都不是單一使用,RIP-seq也不例外,它會搭配其他組學一起對研究的深度和廣度進行擴充。RIP-seq可以與多種技術聯合分析,RNA-seq、m6A-seq等。
案例分析1:m6A識別蛋白YTHDF1在低氧適應和非小細胞肺癌發生發展中的重要功能
YTHDF1 links hypoxia adaptation and non-small cell lung cancer progression. Nat Commun 10, 4892 (2019)..
研究內容:研究發現YTHDF1作為m6A修飾后的RNA結合蛋白家族成員之一,在高原家養哺乳動物中低表達,而在正常肺上皮細胞中抑制其表達可以抵抗低氧誘導的細胞凋亡。深入研究發現,YTHDF1在非小細胞肺癌腫瘤組織和細胞系中均高表達,其在常氧條件下通過加速CDK2和CDK4等細胞周期蛋白的表達來促進腫瘤細胞的增殖;而在鉑類藥物為主的化療壓力環境下,由于YTHDF1高表達促進了Keap1蛋白的表達,導致Nrf2轉錄因子的迅速降解和下游耐藥基因AKR1C1的沉默,因此腫瘤患者對化療更敏感且總生存時間更長
研究技術:RIP-seq、m6A-seq、RNA-seq
研究總結:聯合運用了RIP-seq和m6A-seq,鑒定出細胞周期因子CDK2和CDK4的m6A修飾發生了變化,且被YTHDF1所識別和結合。CDK2和CDK4表達的紊亂也導致細胞周期的紊亂,使得癌細胞增殖加速

圖1 m6A-seq和 RIP-seq揭示YTHDF1調控模型

圖2 IGV圖展示CDK2和CDK4的RNA結合蛋白富集差異和m6A修飾差異
案例內容
結果內容
建議內容
在線留言
相關產品
 選擇區號?
選擇區號?復制成功
×


 瀏覽器自帶分享功能也很好用哦~
瀏覽器自帶分享功能也很好用哦~

 瀏覽器自帶分享功能也很好用哦~
瀏覽器自帶分享功能也很好用哦~